📕문제
DNA란 어떤 유전물질을 구성하는 분자이다. 이 DNA는 서로 다른 4가지의 뉴클레오티드로 이루어져 있다(Adenine, Thymine, Guanine, Cytosine). 우리는 어떤 DNA의 물질을 표현할 때, 이 DNA를 이루는 뉴클레오티드의 첫 글자를 따서 표현한다. 만약에 Thymine-Adenine-Adenine-Cytosine-Thymine-Guanine-Cytosine-Cytosine-Guanine-Adenine-Thymine로 이루어진 DNA가 있다고 하면, “TAACTGCCGAT”로 표현할 수 있다. 그리고 Hamming Distance란 길이가 같은 두 DNA가 있을 때, 각 위치의 뉴클오티드 문자가 다른 것의 개수이다. 만약에 “AGCAT"와 ”GGAAT"는 첫 번째 글자와 세 번째 글자가 다르므로 Hamming Distance는 2이다.
우리가 할 일은 다음과 같다. N개의 길이 M인 DNA s1, s2, ..., sn가 주어져 있을 때 Hamming Distance의 합이 가장 작은 DNA s를 구하는 것이다. 즉, s와 s1의 Hamming Distance + s와 s2의 Hamming Distance + s와 s3의 Hamming Distance... 의 합이 최소가 된다는 의미이다.
📕입력
첫 줄에 DNA의 수 N과 문자열의 길이 M이 주어진다. 그리고 둘째 줄부터 N+1번째 줄까지 N개의 DNA가 주어진다. N은 1,000보다 작거나 같은 자연수이고, M은 50보다 작거나 같은 자연수이다.
📕출력
첫째 줄에 Hamming Distance의 합이 가장 작은 DNA 를 출력하고, 둘째 줄에는 그 Hamming Distance의 합을 출력하시오. 그러한 DNA가 여러 개 있을 때에는 사전 순으로 가장 앞서는 것을 출력한다.
💡문제해석
굉장히 문제 해석하기 당황스러운 문제였다;;,,
처음에 난이도가 실버 5길래 굉장히 쉽게 풀릴 줄 알았는데 문제를 이해하는데서 좀 골이 아팠다.
여기서 등장하는 Hamming Distance라는 단어가 등장하는데 내 글에서는 간략하게 HD라고 설명하겠다.
HD가 최소가 되는 DNA가 무슨 말인지 한참을 걸려서 이해했다.
처음에 나는 주어진 문자열 중에서 가장 차이가 적은 문자열을 뽑았는데 답과 틀려서 당황했다,;;
문제가 요구하는 것은 입력에서 주어지는 N개의 DNA 서열과 비교해서 임의의 DNA를 만들고 그 DNA후보 중에서
가장 HD가 작은 염기서열을 내가 만드는 것이다.
👀HD가 작은 염기서열은 각 자릿수 별로 가장 많이 등장하는 알파벳으로 구성하면 된다. 그래야 주어진 DNA들과의 HD가 최소가 된다.
구현
1. 변수 선언
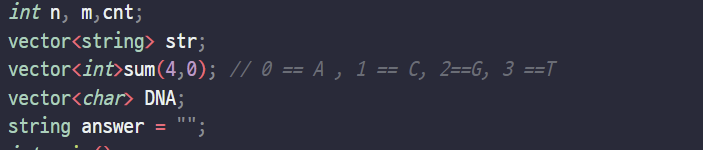
- n과 m은 n개의 DNA의 종류의 개수를 의미하고 m은 주어진 DNA에서 문자의 개수를 의미한다.
- str은 DNA 서열을 저장하기 위한 vector
- sum은 각 알파벳(염기서열)의 빈도수를 측정하기 위한 저장 vector
- 빈도수가 같을 시 오름차순으로 정렬해야 하기 때문에 나는 편의상 알파벳이 작은 순서대로 index를 할당했다.
- DNA vector는 이제 sum에 해당하는 index에 알파벳을 저장했다.
- answer는 내가 최종적으로 구할 DNA를 출력
2. 빈도수 측정

- 나는 n개의 DNA가 있을 때 각 자릿수 별로 알파벳의 빈도수를 측정했다.
- 각자가 대응되는 sum을 증가시켰다.
3. 알파벳 정하기
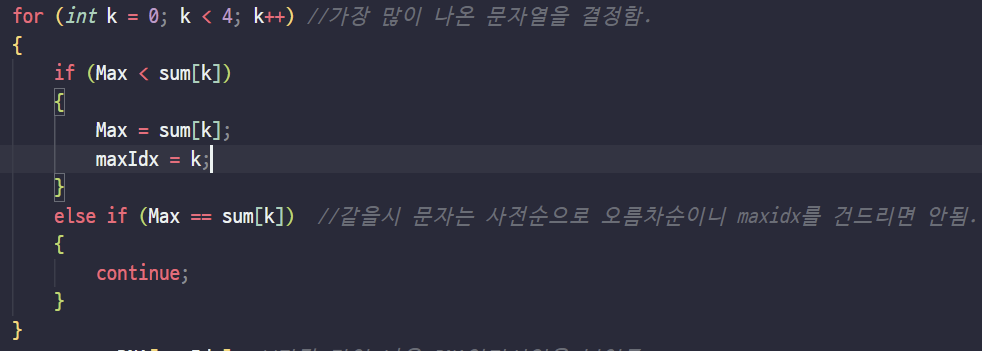
- 최종적으로 HD가 작은 서열을 만들기 위해선 각 자리에서 빈도수가 높은 알파벳을 선정해서 넣어주면 된다.
- 여기서 만약 빈도수가 같을 시 자동적으로 내가 오름차순으로 정렬되게 만들었다.(위의 변수 선언에 자세한 설명이 있음.)
4.HD의 값 계산

📜코드
/* DNA 1969 */
#include <iostream>
#include <vector>
#include <algorithm>
using namespace std;
int n, m,cnt;
vector<string> str;
vector<int>sum(4,0); // 0 == A , 1 == C, 2==G, 3 ==T
vector<char> DNA;
string answer = "";
int main()
{
cin >> n >> m;
DNA.push_back('A'),DNA.push_back('C'),DNA.push_back('G'),DNA.push_back('T');
for (int i = 0; i < n; i++)
{
string s;
cin >> s;
str.push_back(s);
}
for (int i = 0; i < m; i++)
{
int Max=0,maxIdx=0;
for (int j = 0; j < n; j++) //각 DNA의 자리수별로 등장하는 염기서열의 빈도수를 sum에다 집어넣음.
{
if (str[j][i] == 'A')
{
sum[0]++;
}
else if (str[j][i] == 'C')
{
sum[1]++;
}
else if (str[j][i] == 'G')
{
sum[2]++;
}
else if (str[j][i] == 'T')
{
sum[3]++;
}
}
// cout<<endl;
for (int k = 0; k < 4; k++) //가장 많이 나온 문자열을 결정함.
{
if (Max < sum[k])
{
Max = sum[k];
maxIdx = k;
}
else if (Max == sum[k]) //같을시 문자는 사전순으로 오름차순이니 maxidx를 건드리면 안됨. 왜냐하면 내가 sum에다 넣을때 이미 오름차순으로 넣음.
{
continue;
}
}
answer += DNA[maxIdx]; //가장 많이 나온 DNA염기서열을 넣어줌.
sum.assign(4,0); //빈도수 초기화
}
cout<<answer<<endl;
for(int i=0; i<n;i++)
{
for(int j=0; j<m;j++)
{
if(answer[j]!=str[i][j]) //내가 찾은 염기서열과 다른 염기서열을 비교해서 다른 글자수가 있다면 ++해줌.
{
cnt++;
}
}
}
cout<<cnt;
}✔느낀 점
- 조금 더 간결하고 효율적으로 풀 수 있을 것 같지만, 처음 생각나는 대로 문제를 해결했다.
- switch문을 사용해도 될 거 같고, pair를 사용해도 될거같고, 하지만 정말 무식하게 풀긴 했다.(다시 보니까 ㅎㅎ..)
- 문제만 잘 이해하면 쉽게 풀 수 있는 문제이다.
- 핵심은 HD가 최소인 서열을 만드는 것!
- 알고리즘적으로는 전혀 어려운 문제는 아니다.
'CodingTest > Baekjoon' 카테고리의 다른 글
| [백준 11047] 동전0 (C++) (0) | 2022.07.22 |
|---|---|
| [백준 1339] 단어수학(C++) (0) | 2022.07.22 |
| [백준 2800] 괄호제거(C++) (0) | 2022.07.20 |
| [백준 17140] 이차원 배열과 연산(C++) (0) | 2022.07.20 |
| [백준 9935]-문자열폭발(C++) (0) | 2022.07.19 |
📕문제
DNA란 어떤 유전물질을 구성하는 분자이다. 이 DNA는 서로 다른 4가지의 뉴클레오티드로 이루어져 있다(Adenine, Thymine, Guanine, Cytosine). 우리는 어떤 DNA의 물질을 표현할 때, 이 DNA를 이루는 뉴클레오티드의 첫 글자를 따서 표현한다. 만약에 Thymine-Adenine-Adenine-Cytosine-Thymine-Guanine-Cytosine-Cytosine-Guanine-Adenine-Thymine로 이루어진 DNA가 있다고 하면, “TAACTGCCGAT”로 표현할 수 있다. 그리고 Hamming Distance란 길이가 같은 두 DNA가 있을 때, 각 위치의 뉴클오티드 문자가 다른 것의 개수이다. 만약에 “AGCAT"와 ”GGAAT"는 첫 번째 글자와 세 번째 글자가 다르므로 Hamming Distance는 2이다.
우리가 할 일은 다음과 같다. N개의 길이 M인 DNA s1, s2, ..., sn가 주어져 있을 때 Hamming Distance의 합이 가장 작은 DNA s를 구하는 것이다. 즉, s와 s1의 Hamming Distance + s와 s2의 Hamming Distance + s와 s3의 Hamming Distance... 의 합이 최소가 된다는 의미이다.
📕입력
첫 줄에 DNA의 수 N과 문자열의 길이 M이 주어진다. 그리고 둘째 줄부터 N+1번째 줄까지 N개의 DNA가 주어진다. N은 1,000보다 작거나 같은 자연수이고, M은 50보다 작거나 같은 자연수이다.
📕출력
첫째 줄에 Hamming Distance의 합이 가장 작은 DNA 를 출력하고, 둘째 줄에는 그 Hamming Distance의 합을 출력하시오. 그러한 DNA가 여러 개 있을 때에는 사전 순으로 가장 앞서는 것을 출력한다.
💡문제해석
굉장히 문제 해석하기 당황스러운 문제였다;;,,
처음에 난이도가 실버 5길래 굉장히 쉽게 풀릴 줄 알았는데 문제를 이해하는데서 좀 골이 아팠다.
여기서 등장하는 Hamming Distance라는 단어가 등장하는데 내 글에서는 간략하게 HD라고 설명하겠다.
HD가 최소가 되는 DNA가 무슨 말인지 한참을 걸려서 이해했다.
처음에 나는 주어진 문자열 중에서 가장 차이가 적은 문자열을 뽑았는데 답과 틀려서 당황했다,;;
문제가 요구하는 것은 입력에서 주어지는 N개의 DNA 서열과 비교해서 임의의 DNA를 만들고 그 DNA후보 중에서
가장 HD가 작은 염기서열을 내가 만드는 것이다.
👀HD가 작은 염기서열은 각 자릿수 별로 가장 많이 등장하는 알파벳으로 구성하면 된다. 그래야 주어진 DNA들과의 HD가 최소가 된다.
구현
1. 변수 선언
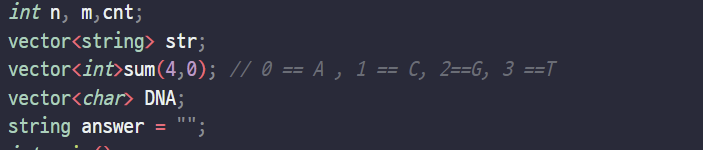
- n과 m은 n개의 DNA의 종류의 개수를 의미하고 m은 주어진 DNA에서 문자의 개수를 의미한다.
- str은 DNA 서열을 저장하기 위한 vector
- sum은 각 알파벳(염기서열)의 빈도수를 측정하기 위한 저장 vector
- 빈도수가 같을 시 오름차순으로 정렬해야 하기 때문에 나는 편의상 알파벳이 작은 순서대로 index를 할당했다.
- DNA vector는 이제 sum에 해당하는 index에 알파벳을 저장했다.
- answer는 내가 최종적으로 구할 DNA를 출력
2. 빈도수 측정

- 나는 n개의 DNA가 있을 때 각 자릿수 별로 알파벳의 빈도수를 측정했다.
- 각자가 대응되는 sum을 증가시켰다.
3. 알파벳 정하기

- 최종적으로 HD가 작은 서열을 만들기 위해선 각 자리에서 빈도수가 높은 알파벳을 선정해서 넣어주면 된다.
- 여기서 만약 빈도수가 같을 시 자동적으로 내가 오름차순으로 정렬되게 만들었다.(위의 변수 선언에 자세한 설명이 있음.)
4.HD의 값 계산

📜코드
/* DNA 1969 */ #include <iostream> #include <vector> #include <algorithm> using namespace std; int n, m,cnt; vector<string> str; vector<int>sum(4,0); // 0 == A , 1 == C, 2==G, 3 ==T vector<char> DNA; string answer = ""; int main() { cin >> n >> m; DNA.push_back('A'),DNA.push_back('C'),DNA.push_back('G'),DNA.push_back('T'); for (int i = 0; i < n; i++) { string s; cin >> s; str.push_back(s); } for (int i = 0; i < m; i++) { int Max=0,maxIdx=0; for (int j = 0; j < n; j++) //각 DNA의 자리수별로 등장하는 염기서열의 빈도수를 sum에다 집어넣음. { if (str[j][i] == 'A') { sum[0]++; } else if (str[j][i] == 'C') { sum[1]++; } else if (str[j][i] == 'G') { sum[2]++; } else if (str[j][i] == 'T') { sum[3]++; } } // cout<<endl; for (int k = 0; k < 4; k++) //가장 많이 나온 문자열을 결정함. { if (Max < sum[k]) { Max = sum[k]; maxIdx = k; } else if (Max == sum[k]) //같을시 문자는 사전순으로 오름차순이니 maxidx를 건드리면 안됨. 왜냐하면 내가 sum에다 넣을때 이미 오름차순으로 넣음. { continue; } } answer += DNA[maxIdx]; //가장 많이 나온 DNA염기서열을 넣어줌. sum.assign(4,0); //빈도수 초기화 } cout<<answer<<endl; for(int i=0; i<n;i++) { for(int j=0; j<m;j++) { if(answer[j]!=str[i][j]) //내가 찾은 염기서열과 다른 염기서열을 비교해서 다른 글자수가 있다면 ++해줌. { cnt++; } } } cout<<cnt; }
✔느낀 점
- 조금 더 간결하고 효율적으로 풀 수 있을 것 같지만, 처음 생각나는 대로 문제를 해결했다.
- switch문을 사용해도 될 거 같고, pair를 사용해도 될거같고, 하지만 정말 무식하게 풀긴 했다.(다시 보니까 ㅎㅎ..)
- 문제만 잘 이해하면 쉽게 풀 수 있는 문제이다.
- 핵심은 HD가 최소인 서열을 만드는 것!
- 알고리즘적으로는 전혀 어려운 문제는 아니다.
'CodingTest > Baekjoon' 카테고리의 다른 글
| [백준 11047] 동전0 (C++) (0) | 2022.07.22 |
|---|---|
| [백준 1339] 단어수학(C++) (0) | 2022.07.22 |
| [백준 2800] 괄호제거(C++) (0) | 2022.07.20 |
| [백준 17140] 이차원 배열과 연산(C++) (0) | 2022.07.20 |
| [백준 9935]-문자열폭발(C++) (0) | 2022.07.19 |
